Manipulación avanzada de dataframes
Funciones y operadores
En la lección autónoma Manipulación básica de dataframes hemos empleando la librería base y los operadores más clásicos para manipular conjuntos de datos basados en sus índices. Vamos ahora a ahondar en la manipulación de dataframes con funciones de más alto nivel.
Trabajaremos con las funciones que nos permiten tratar valores faltantes, ampliar un dataframe, por filas o columnas: rbind(), cbind(), merge() y nos centraremos en funciones más allá de los operadores basados en índices del data.frame como son las funciones: aggregate() y la familia de funciones apply.
Limpieza del dataframe con funciones base
Cambiar los nombres de filas y columnas
Los dataframes también pueden tener un atributo names que indica los nombres de sus columnas (si los tiene). En general, entendemos las columnas como variables
names(mtcars)## [1] "mpg" "cyl" "disp" "hp" "drat" "wt" "qsec" "vs" "am" "gear"
## [11] "carb"Es un vector de caracteres que incluye los nombres del conjunto de datos mtcars.
Este vector es modificable. Cambia el nombre de la segunda columna por su equivalente en español (cilindrada).
names(mtcars)...names(mtcars)[2] <- "cilindrada"De la misma naturaleza son los nombres de las columnas:
rownames(mtcars)## [1] "Mazda RX4" "Mazda RX4 Wag" "Datsun 710"
## [4] "Hornet 4 Drive" "Hornet Sportabout" "Valiant"
## [7] "Duster 360" "Merc 240D" "Merc 230"
## [10] "Merc 280" "Merc 280C" "Merc 450SE"
## [13] "Merc 450SL" "Merc 450SLC" "Cadillac Fleetwood"
## [16] "Lincoln Continental" "Chrysler Imperial" "Fiat 128"
## [19] "Honda Civic" "Toyota Corolla" "Toyota Corona"
## [22] "Dodge Challenger" "AMC Javelin" "Camaro Z28"
## [25] "Pontiac Firebird" "Fiat X1-9" "Porsche 914-2"
## [28] "Lotus Europa" "Ford Pantera L" "Ferrari Dino"
## [31] "Maserati Bora" "Volvo 142E"Valores faltantes
¿Cómo tratamos a los valores faltantes? Son uno de los quebraderos de cabeza de los investigadores ya que pueden alterar el resultado de los experimentos. R nos apoya en la tarea de detectarlos, inputarlos o borrarlos con las siguientes funciones.
Para comprobar si hay valores faltantes se puede utilizar la (a estas alturas ya famosa) función summary().
coches <- read.table("http://gauss.inf.um.es/datos/coches.csv", sep=";", head=T)
summary(coches)¿Cómo podemos combinar las funciones colSums() y is.na() para obtener una tabla que indique el número de valores faltantes por columna?
... is.na(coches) ..colSums(is.na(coches))Utiliza las funciones which() y is.na() para encontrar las filas en las que la varible hp tiene los valores faltantes.
...which(is.na(coches$hp))Si queremos imputar los valores faltantes con un valor fijo solo tenemos que seleccionarlos y asignar dicho valor a la selección como sigue:
cochesI <- coches
cochesI[is.na(cochesI)] <- 10Calcula la mediana de la columna 2 (cyl o cilindrada) e imputa los valores faltantes de esa columna con ella.
mediana <- median(...)
... <- medianamediana <- median(coches$cilindrada, na.rm=T)
coches$cilindrada[is.na(coches$cilindrada)] <- mediana
colSums(is.na(coches)) # esta parte no es necesaria, solo para comprobar que se han imputadoLa función complete.cases() devuelve un valor lógico indicando qué filas del conjunto de datos están completas, es decir, no tienen valores faltantes.
complete.cases(coches)## [1] FALSE TRUE TRUE TRUE FALSE TRUE TRUE FALSE TRUE TRUE TRUE TRUE
## [13] TRUE FALSE TRUE TRUE FALSE TRUE TRUE FALSE TRUE TRUE TRUE TRUE
## [25] TRUE TRUE TRUE TRUE TRUE FALSE TRUE TRUEUtiliza la función complete.cases() para crear un nuevo dataframe, basado en coches pero que se haya deshecho de las filas donde hubiera algún valor faltante. Comprueba que ya no hay ningún valor faltante en el nuevo dataframe.
cochesLimpio <- ...complete.cases(coches)..
...cochesLimpio <- coches[complete.cases(coches),]
colSums(is.na(cochesLimpio))Combinar dataframes
Ahora vamos a aprender a unir dataframes. Trabajaremos con estos conjuntos de datos:
paises1 <- read.table("http://gauss.inf.um.es/datos/countries_sub1.csv", sep=";", head=T)
paises2 <- read.table("http://gauss.inf.um.es/datos/countries_sub2.csv", sep=";", head=T)
paises3 <- read.table("http://gauss.inf.um.es/datos/countries_sub3.csv", sep=";", head=T)
paises4 <- read.table("http://gauss.inf.um.es/datos/countries_sub4.csv", sep=";", head=T)| Country | MigracionNeta |
|---|---|
| Argentina | 0.61 |
| Alemania | 2.18 |
| Country | MigracionNeta |
|---|---|
| Rusia | 1.00 |
| España | 0.99 |
| Grecia | 2.35 |
| Pais | MigracionNeta |
|---|---|
| Grecia | 2.35 |
| Honduras | -1.99 |
| Country | RatioNacimientos |
|---|---|
| España | 10.06 |
| Argentina | 16.73 |
| Rusia | 9.95 |
Cuando trabajamos con datos que proceden de diversas fuentes, nos puede interesar unirlos. Los datos a unir deben tener una estructura común: mismas variables (incluyendo nombre y tipo) o misma longitud.
- Añadir más filas:
rbind()(row bind) sirve para anexar las filas de 2 dataframes.
paises <- rbind(paises1, paises2)| Country | MigracionNeta |
|---|---|
| Argentina | 0.61 |
| Alemania | 2.18 |
| Rusia | 1.00 |
| España | 0.99 |
| Grecia | 2.35 |
Une ahora el conjunto anterior paises con paises3. ¿Qué sucede? ¿Puedes solucionarlo?
# Si hacemos rbind(paises, paises3) los nombres de las columnas no son los mismos
names(paises3)[1] <- "Country" # o aún mejor: names(paises3)[1] <- names(paises)[1]
rbind(paises, paises3)- Añadir más columnas
cbind()(column bind) ymerge()sirven para anexar las columnas de 2 dataframes que tienen las mismas variables.
paises5 <- cbind(paises2, paises4)| Country | MigracionNeta | Country | RatioNacimientos |
|---|---|---|---|
| Rusia | 1.00 | España | 10.06 |
| España | 0.99 | Argentina | 16.73 |
| Grecia | 2.35 | Rusia | 9.95 |

En este caso, cbind devuelve una tabla que no sigue los principios tidy ya que la variable Country aparece repetida.
Para evitar este problema al combinar 2 dataframes se utiliza la función merge(). Esta función cuenta con un argumento by donde se indica la variable (o clave) por la que unir los dataframes.
En este caso, la clave por la que se desea unir los dataframes es la variable Country y los dataframes solo tienen dos sujetos en común: España y Rusia.
merge(paises2,paises4,by="Country")| Country | MigracionNeta | RatioNacimientos |
|---|---|---|
| España | 0.99 | 10.06 |
| Rusia | 1.00 | 9.95 |
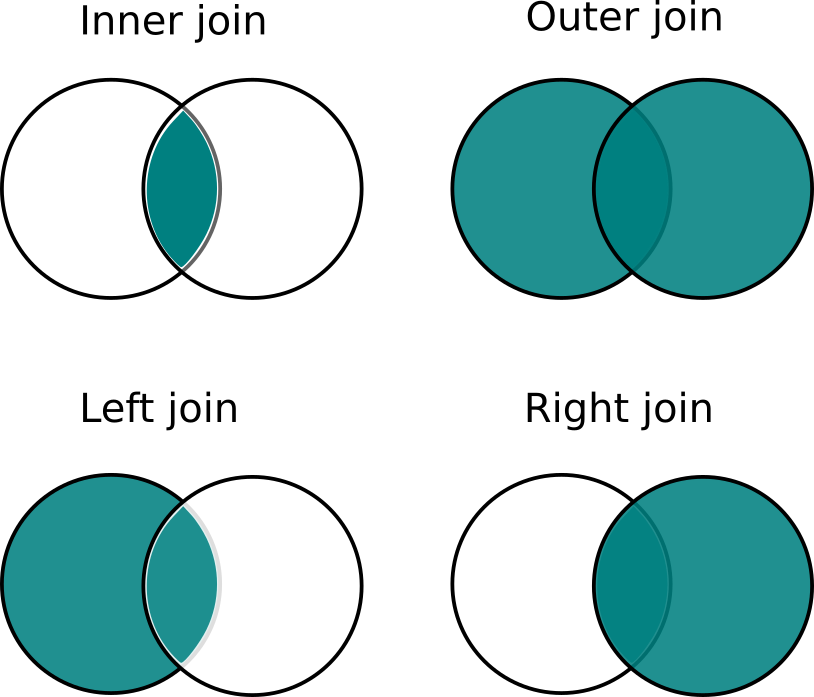
| Inner Join | merge(df1, df2, by='clave') | solo sujetos comunes |
| Outer Join | merge(df1, df2, by='clave', all=TRUE) | todos los sujetos |
| Left outer | merge(df1, df2, by='clave', all.x=TRUE) | todos del izdo. y comunes con el dcho. |
| Rigth outer | merge(df1, df2, by='clave', all.y=TRUE) | todos del dcho. y comunes con el izdo. |
Recordamos que
| Country | MigracionNeta |
|---|---|
| Rusia | 1.00 |
| España | 0.99 |
| Grecia | 2.35 |
| Country | RatioNacimientos |
|---|---|
| España | 10.06 |
| Argentina | 16.73 |
| Rusia | 9.95 |
Utiliza R o deduce las siguientes preguntas:
| Country | MigracionNeta | RatioNacimientos |
|---|---|---|
| Argentina | NA | 16.73 |
| España | 0.99 | 10.06 |
| Grecia | 2.35 | NA |
| Rusia | 1.00 | 9.95 |
| Country | MigracionNeta | RatioNacimientos |
|---|---|---|
| Argentina | NA | 16.73 |
| España | 0.99 | 10.06 |
| Rusia | 1.00 | 9.95 |
| Country | MigracionNeta | RatioNacimientos |
|---|---|---|
| España | 0.99 | 10.06 |
| Grecia | 2.35 | NA |
| Rusia | 1.00 | 9.95 |
Aggregate
La palabra agregar tiene varios significados, entre los que se incluye añadir unas cosas o personas a otras del mismo tipo o unir varias cosas similares.
Aggregate() es una función base de R que sirve para agregar un dataframe utilizando una función especificada en el argumento FUN en las columnas del dataframe definidas por el argumento by . El argumento by tiene que ser una lista.
Por ejemplo, el conjunto de datos mtcars tiene una columna dedicada a la cilindrada (cyl) que toma los valores del conjunto {4,6,8}.
| mpg | cyl | disp | hp | drat | wt | qsec | vs | am | gear | carb | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Mazda RX4 | 21.0 | 6 | 160.0 | 110 | 3.90 | 2.620 | 16.46 | 0 | 1 | 4 | 4 |
| Mazda RX4 Wag | 21.0 | 6 | 160.0 | 110 | 3.90 | 2.875 | 17.02 | 0 | 1 | 4 | 4 |
| Datsun 710 | 22.8 | 4 | 108.0 | 93 | 3.85 | 2.320 | 18.61 | 1 | 1 | 4 | 1 |
| Hornet 4 Drive | 21.4 | 6 | 258.0 | 110 | 3.08 | 3.215 | 19.44 | 1 | 0 | 3 | 1 |
| Hornet Sportabout | 18.7 | 8 | 360.0 | 175 | 3.15 | 3.440 | 17.02 | 0 | 0 | 3 | 2 |
| Valiant | 18.1 | 6 | 225.0 | 105 | 2.76 | 3.460 | 20.22 | 1 | 0 | 3 | 1 |
| Duster 360 | 14.3 | 8 | 360.0 | 245 | 3.21 | 3.570 | 15.84 | 0 | 0 | 3 | 4 |
| Merc 240D | 24.4 | 4 | 146.7 | 62 | 3.69 | 3.190 | 20.00 | 1 | 0 | 4 | 2 |
Vamos a utilizar la función aggregate() para calcular la media de todas las variables del dataset mtcars para cada uno de los valores de la variable cyl. Para ellos los argumentos son:
mtcars: el conjunto de datosby = list(cyl): una lista con el nombre de la variable por la que queremos aplicar la funciónFUN = mean: la función media
aggMtcars <-aggregate(mtcars, by = list(mtcars$cyl), FUN = mean)
aggMtcarsEn la tabla anterior se muestran las medias para todas las columnas de mtcars agrupándolas por cyl.
A continuaciĺon, realizaremos ejercicios en los que se agregarán distintas columnas del conjunto de datos CO2. Una vista reducida del conjunto es la siguiente:
| Plant | Type | Treatment | conc | uptake | |
|---|---|---|---|---|---|
| 1 | Qn1 | Quebec | nonchilled | 95 | 16.0 |
| 2 | Qn1 | Quebec | nonchilled | 175 | 30.4 |
| 22 | Qc1 | Quebec | chilled | 95 | 14.2 |
| 23 | Qc1 | Quebec | chilled | 175 | 24.1 |
| 43 | Mn1 | Mississippi | nonchilled | 95 | 10.6 |
| 44 | Mn1 | Mississippi | nonchilled | 175 | 19.2 |
| 64 | Mc1 | Mississippi | chilled | 95 | 10.5 |
| 65 | Mc1 | Mississippi | chilled | 175 | 14.9 |
Calcula la media de absorción (uptake) por origen de planta (Type) usando aggregate(). Completa la función
aggregate(.., by = list(..), FUN=mean)aggregate(CO2$uptake, by = list(CO2$Type), FUN=mean)Ahora calcula el máximo de absorción (uptake) por origen de planta y por planta (Type, Plant) usando aggregate().
aggregate(.., .., ..)aggregate(CO2$uptake, by = list(CO2$Type, CO2$Plant), FUN=max)Vamos a construir una tabla de estadísticos descriptivos personalizada. Nos interesa obtener la suma, media y desviación estándar de absorción (uptake) por tipo de planta (Type).
Ya sabemos qué va en los dos primeros argumentos pero, ¿cómo es la función? Debemos construirla nosotras mismas:
NOTA: Las soluciones de los dos ejercicios siguientes no se imprimen correctamente en el curso online por razones que escapan a nuestro entendimiento, rogamos lo pruebes en la consola normal de R para ver el resultado.
f.custom <- function(x){
c(Suma = ...,
Media = ...,
SD = ...)
}
aggregate(CO2$uptake, by = list(CO2$Type), FUN = f.custom)f.custom <- function(x){
c(Suma = sum(x),
Media = mean(x),
SD = sd(x))
}
as.data.frame(aggregate(CO2$uptake, by = list(CO2$Type), FUN = f.custom))Añade dos columnas más que sean el máximo y la mediana y pon otro nombre a la función
... <- function(x){
c(Suma = ...,
Media = ...,
SD = ...,
...,
...)
}
aggregate(CO2$uptake, by = list(CO2$Type), FUN = )descriptivos <- function(x){
c(Suma = sum(x),
Media = mean(x),
SD = sd(x),
MAX = max(x),
MD = median(x))
}
aggregate(CO2$uptake, by = list(CO2$Type), FUN = descriptivos)Los valores de entrada en aggregate se pueden especificar también como una fórmula.
De forma básica, las fórmulas en R relacionan conceptos usando la virgulilla ~. A la izquierda se encuentra la variable independiente y a la derecha la o las dependientes que se relacionan usando el signo más.
Veamos un ejemplo de cómo usar la fórmula en aggregate.
aggregate(CO2$uptake, by = list(CO2$Type, CO2$Plant), FUN=max)aggregate(uptake ~ Type+Plant, data=CO2, FUN=max)Con esta estructura, evitas llamar al dataframe cada vez que invocas una variable.
Las fórmulas no se pueden usar como entrada de todas las funciones de R, pero son parte fundamental de este lenguaje.
Algunos componentes de la familia Apply: apply(), lapply() y tapply()
En este módulo aprenderás sobre el uso de la función apply en R y algunas variantes en los conjuntos de datos.
La familia apply()
La familia apply() pertenece al paquete base de R y se utiliza para manipular porciones de los datos de una forma repetitiva. Estas funciones permiten cruzar los datos de diversas formas evitando utilizar bucles, que son menos eficientes y el código es más engorroso.
Si no sabes lo que son los bucles en programación, no son necesarios para seguir el curso pero quizás eres curios@. En ese caso puedes completar la información con https://www.datacamp.com/community/tutorials/tutorial-on-loops-in-r.
La familia incluye las fuciones apply(), lapply() , sapply(), vapply(), mapply(), rapply(), y tapply().
¿Cuándo y cómo utilizar cada una de ellas?
Depende de la estructura de los datos que se quieran manipular y del formato de salida que se necesite.
apply
apply() opera en vectores y matrices. Sus argumentos básicos son apply(X, MARGIN, FUN)
donde
- X es un vector o una matriz
MARGIN es una variable que define cómo se aplica la función:
- MARGIN = 1 --> por filas - MARGIN = 2 --> por columnas - MARGIN = c(1,2) por filas y columnasFUN es la función que se desea aplicar a los datos
Como todas las filas o todas las columnas se van a ver afectadas por la misma función, éstas tienen que ser del mismo tipo. Para ello crearemos un dataframe muy simple:
df<- matrix(c(1:10, 1:10, 1:10), nrow = 10, ncol = 3)
df## [,1] [,2] [,3]
## [1,] 1 1 1
## [2,] 2 2 2
## [3,] 3 3 3
## [4,] 4 4 4
## [5,] 5 5 5
## [6,] 6 6 6
## [7,] 7 7 7
## [8,] 8 8 8
## [9,] 9 9 9
## [10,] 10 10 10Y, a continuación mostramos la suma por filas y por columnas respectivamente utilizando la función apply
apply(df, 1, sum)## [1] 3 6 9 12 15 18 21 24 27 30apply(df, 2, sum)## [1] 55 55 55La función sum ya existe en R, pero en algunas ocasiones será útil definir nuestrar propias funciones. Utiliza la función st.err definida a continuación para calcular el error estándard por columnas de df:
st.err <- function(x){
sd(x)/sqrt(length(x))
}
apply(df, ... , ...)st.err <- function(x){
sd(x)/sqrt(length(x))
}
apply(df,2, st.err)Utiliza la función apply para calcular el rango de las variables numéricas del dataset CO2
apply (CO2[...], ..., range)apply (CO2[ , 4:5], 2, range)lapply()
Veamos el funcionamiento de la función lapply() con este simple ejemplo:
lapply(1:3, function(x) x^2)## [[1]]
## [1] 1
##
## [[2]]
## [1] 4
##
## [[3]]
## [1] 9Lapply devuelve una lista de la misma longitud de su argumento de entrada.
Los dataframes son listas de vectores del mismo tamaño, por lo tanto, lapply() resulta muy útil para aplicar para aplicar una función a las columnas de un dataframe.
El conjunto de datos starwarsjuguetes presenta la siguiente codificación:
| genero | 1:femenino, 2:masculino |
| jedi | 1:jedi, 2:no jedi |
| especie | 1:humano, 2:droide, 3:yoda, 4:wookiee |
| arma | 1:dinamita, 2:rifleBayesta, 3:sable de luz, 4:sinArmas |
starwars <- read.table("starwarsjuguetes.csv", sep=";", head=T)
summary(starwars)## nombre genero altura peso
## Length:8 Min. :1.000 Min. :0.660 Min. : 17.00
## Class :character 1st Qu.:2.000 1st Qu.:1.365 1st Qu.: 44.75
## Mode :character Median :2.000 Median :1.695 Median : 76.00
## Mean :1.875 Mean :1.551 Mean : 64.88
## 3rd Qu.:2.000 3rd Qu.:1.805 3rd Qu.: 77.75
## Max. :2.000 Max. :2.280 Max. :112.00
## jedi especie arma
## Min. :1.000 Min. :1.000 Min. :1.000
## 1st Qu.:1.000 1st Qu.:1.000 1st Qu.:1.750
## Median :2.000 Median :1.500 Median :3.000
## Mean :1.625 Mean :1.875 Mean :2.625
## 3rd Qu.:2.000 3rd Qu.:2.250 3rd Qu.:3.250
## Max. :2.000 Max. :4.000 Max. :4.000Las variables género, jedi, especie y arma son categóricas, sin embargo, se tratan como numéricas ya que no lo hemos especificado. Además de convertirlas de forma individual en factores, podemos hacerlo de forma más inteligente, esto es, utilizando la función lapply() en las columnas seleccionadas:
starwars[c(2,5:7)] <- lapply(starwars[c(2,5:7)], as.factor)
summary(starwars)## nombre genero altura peso jedi especie arma
## Length:8 1:1 Min. :0.660 Min. : 17.00 1:3 1:4 1:2
## Class :character 2:7 1st Qu.:1.365 1st Qu.: 44.75 2:5 2:2 2:1
## Mode :character Median :1.695 Median : 76.00 3:1 3:3
## Mean :1.551 Mean : 64.88 4:1 4:2
## 3rd Qu.:1.805 3rd Qu.: 77.75
## Max. :2.280 Max. :112.00starwars[c(2,5:7)] <- lapply(starwars[c(2,5:7)], as.factor)Utiliza la función levels() para renombrar los niveles de una de las variables categóricas de acuerdo a la tabla de codificación anterior y muestra un gráfico de barras de ella:
levels()...
barplot(...)levels(starwars$genero) <- c("fem", "masc")
barplot(table(starwars$genero))tapply()
La función tapply es útil cuando necesitamos partir un vector por grupos, realizar cálculos en cada uno de esos grupos y devolver los resultados en forma de tabla. Se pueden utilizar más de un factor para definir los grupos.
tapply(CO2$uptake, CO2$Type, mean)## Quebec Mississippi
## 33.54286 20.88333Diferencias entre aggregate y tapply()
Aggregate está diseñado para aplicar una función a múltiples columnas y devolver un dataframe con una fila por categoría.
Tapply está diseñado para utilizar como entrada un solo vector y los resultados se devuelven en forma de tabla.
aggregate(CO2$uptake, by = list(CO2$Type, CO2$Plant), FUN=max)tapply(CO2$uptake, INDEX=list(CO2$Type, CO2$Plant), max)## Qn1 Qn2 Qn3 Qc1 Qc3 Qc2 Mn3 Mn2 Mn1 Mc2 Mc3 Mc1
## Quebec 39.7 44.3 45.5 38.7 41.4 42.4 NA NA NA NA NA NA
## Mississippi NA NA NA NA NA NA 28.5 32.4 35.5 14.4 19.9 22.2Continuar con tabulaR
Una vez terminado este documento puede:
- Seguir con el siguiente submodulo de tabulaR: Tidyverse: dplyr y tidyr
- Volver al Modulo 1: Manipulación de datos
- Volver al portal de tabulaR